
获得0积分,您同时完成了每日任务,有额外的积分奖励,请前往APP领取
立即前往
原文由 liusky 发表:
是的,从很多脯氨酸的NMR来看,他的同碳上的两氢是不等同的。由于现在做的一些大环肽化合物,其中某些脯氨酸H的位移大大脱离了正常范围,只是猜测是不是一些脯氨酸在这些大环中出现了顺反异构。当然理论上顺反异构是可以从NOESY判断出来的,不过分子过于复杂,TOCSY,NOESY的crossing peaks太多,以前用的500MHz的仪器峰的重叠很严重,现在用700之后可以区分峰的归属,但是判断顺反仍然不足。如果有一些肽中脯氨酸顺反异构的化学位移文献做参考就方便多了。
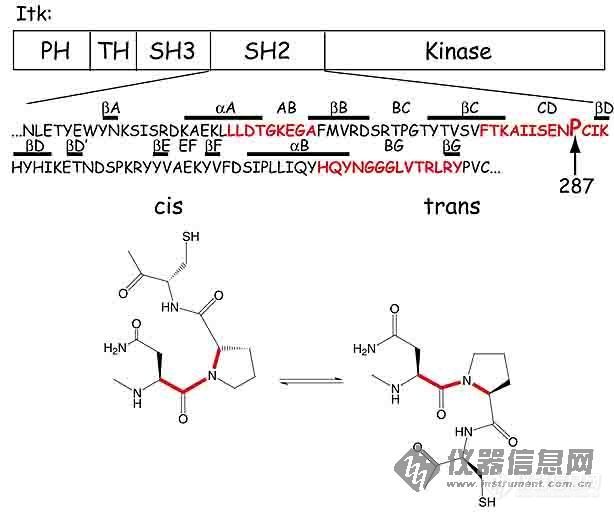 (对不起,发言太少,声望太低,可直接去下面地址看:(http://www.bioscience.org/2005/v10/af/1536/figures.htm)。在蛋白质中多数情况下Pro以trans出现,但在少数情况也会出现cis的,这会导致蛋白质的不同构象,通常都隐含着功能上的不同。这篇文章(http://gepard.bioinformatik.uni-saarland.de/html/Cell_SimulationsSS05/SuggestedReadingV4/V4-Mallis_NSB02.pdf)就是讲的这个问题)。cis和trans会导致不同的构象,当然会影响到相应的化学位移,不单是本身的,更重要的是邻近残基的,如果2种构象同时存在,在15N-1H HSQC中会看到部分2套峰。
(对不起,发言太少,声望太低,可直接去下面地址看:(http://www.bioscience.org/2005/v10/af/1536/figures.htm)。在蛋白质中多数情况下Pro以trans出现,但在少数情况也会出现cis的,这会导致蛋白质的不同构象,通常都隐含着功能上的不同。这篇文章(http://gepard.bioinformatik.uni-saarland.de/html/Cell_SimulationsSS05/SuggestedReadingV4/V4-Mallis_NSB02.pdf)就是讲的这个问题)。cis和trans会导致不同的构象,当然会影响到相应的化学位移,不单是本身的,更重要的是邻近残基的,如果2种构象同时存在,在15N-1H HSQC中会看到部分2套峰。